Untersuchung von Protein-Protein-Interaktionen mit Hilfe der Strep-tag®-Technologie
Das Verständnis von Protein-Protein-Interaktionen (PPI) ist von grundlegender Bedeutung für die Molekularbiologie. Diese Wechselwirkungen regulieren entscheidende biologische Prozesse, von der Signalübertragung über Immunreaktionen bis hin zur Aufrechterhaltung der Zellstruktur und -funktion. Viele Krankheiten sind mit Proteininteraktionen assoziiert. Durch die Untersuchung dieser Interaktionen können Forscher potenzielle therapeutische Ziele ermitteln, Krankheitsmechanismen verstehen, Leads in der Arzneimittelforschung optimieren oder Biomarker für die Diagnose und Überwachung von Krankheiten entdecken.
Die wichtigsten Schritte zur Identifizierung und Charakterisierung der Interaktion zwischen zwei spezifischen Proteinen sind:
- Versuchsplanung: Definition des Zielproteins und Auswahl der geeigneten Methoden basierend auf den spezifischen Zielen und der verfügbaren Ressourcen
- Identifizierung von Leads: Sammeln von Daten, um potenzielle Interaktionspartner zu identifizieren. Dieser Schritt umfasst das Screening und die Identifizierung vielversprechender Kandidaten, die eine signifikante Interaktion zeigen.
- Bestätigung der Interaktion: Validierung der ersten Ergebnisse durch ergänzende Techniken in vitro oder in vivo.
- Charakterisierung: Weitere Charakterisierung der Interaktion in struktureller und funktioneller Hinsicht, z. B. durch Messung der Bindungskinetik
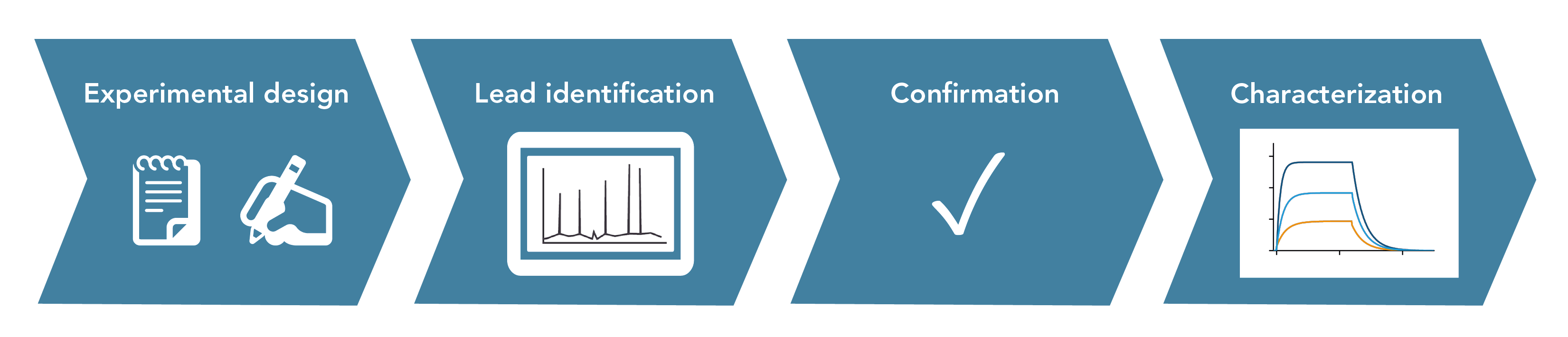
Abbildung 1: Typischer Arbeitsablauf eines Experiments zur Identifizierung und Analyse von Protein-Protein-Interaktionen
Versuchsplanung: Auswahl der Versuchsbedingungen
Um einen einfachen Arbeitsablauf sowie genaue und zuverlässige Ergebnisse zu gewährleisten, sollten bei der Planung des Versuchsaufbaus mehrere Faktoren berücksichtigt werden. Die Auswahl der geeigneten Nachweismethode, des Proteinexpressionssystems und der Bedingungen für die Probenvorbereitung sind entscheidend. Die Verwendung von Protein-Tags, wie Twin-Strep-tag®, His-tag oder FLAG-tag, kann vorteilhaft sein.
Protein-Tags erleichtern die Aufreinigung und den Nachweis von Zielproteinen und können die Effizienz des Experiments verbessern. Sie können auch bei folgenden Schritten hilfreich sein, z. B. bei der Immobilisierung des Proteins während der Analyseschritte, z. B. bei SPR. Um Vorteile für den Analyse Arbeitsablauf zu bieten, sollte der ideale Tag die folgenden Eigenschaften haben:
- keine unspezifische Wechselwirkung
- Hohe Affinität
- Geringer bis kein Einfluss auf die Proteinstruktur
- Geringe Größe
- Umkehrbare Bindung
- Vielfältige Anwendungsmöglichkeiten
Unter den am häufigsten verwendeten Tags besitzt der Twin-Strep-tag® alle diese Eigenschaften und bietet den größten Nutzen für die PPI-Analyse.
Identifizierung von Leads: Affinitätschromatographie, Massenspektrometrie und Bioinformatik
Nachdem ein Zielprotein ausgewählt wurde, müssen die möglichen Interaktionspartner identifiziert werden. Die Isolierung des Zielproteins aus der Zelle unter Beibehaltung seiner Interaktionen ist ein praktischer Weg, dies zu tun. Pull-down-Assays und Co-Immunopräzipitation (Co-IP) sind zwei wirksame Methoden zum Nachweis von Protein-Protein-Wechselwirkungen. Bei der Co-IP werden immobilisierte Antikörper verwendet, um das Bait-Protein zusammen mit dem Prey-Protein aus Zelllysaten zu reinigen, was die Analyse des gesamten Proteinkomplexes ermöglicht. Im Gegensatz dazu wird bei Pull-down-Assays das Zielprotein an eine Affinitätsmarkierung gebunden und zusammen mit allen gebundenen Proteinen mit einem entsprechenden immobilisierten Liganden eingefangen.
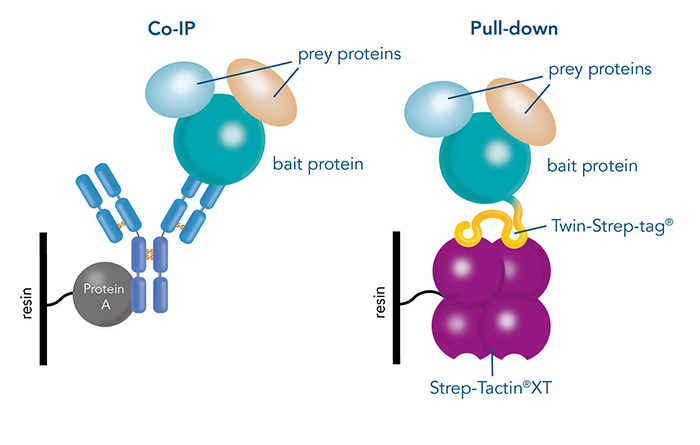
Abbildung 2: Unterschiede zwischen Co-IP mit proteinspezifischen Antikörpern und einem Pull-down, der über einen spezifischen Protein-Tag durchgeführt wird.
Antikörper sind zwar eine gute Wahl, wenn spezifische Antikörper verfügbar sind, doch ist dies nicht immer der Fall. Darüber hinaus können Spezifität und Bindungsaffinität zwischen verschiedenen Antikörpern variieren, was zu unzuverlässigen Ergebnissen führt. Pull-down-Assays, zum Beispiel über Twin-Strep-tag® und Strep-Tactin®XT, bieten mehrere Vorteile. Dasselbe Tag- und Ligandensystem kann für verschiedene Proteine verwendet werden, wobei die gleiche Affinität und Spezifität gewährleistet ist. Außerdem bieten sie milde Elutionsbedingungen, wodurch die Interaktion zwischen den Bait- und Prey-Proteinen erhalten bleibt.
In einem weiteren Schritt werden dann die potenziellen Interaktionspartner identifiziert, zum Beispiel durch Massenspektrometrie. Diese experimentellen Methoden werden durch bioinformatische Methoden ergänzt, die auf der Grundlage von Sequenz- und Strukturdaten aus Datenbanken potenzielle Interaktionspartner vorhersagen. Die Kombination von computergestützten und experimentellen Verfahren erhöht die Genauigkeit und Effizienz der Identifizierung von Leads und ermöglicht es den Forschern, den Pool der zu untersuchenden Kandidaten einzugrenzen. Sobald die vielversprechendsten Leads identifiziert sind, muss jede Interaktion einzeln untersucht und detailliert charakterisiert werden, um die biologische Bedeutung und den Mechanismus der Wechselwirkungen zu verstehen.
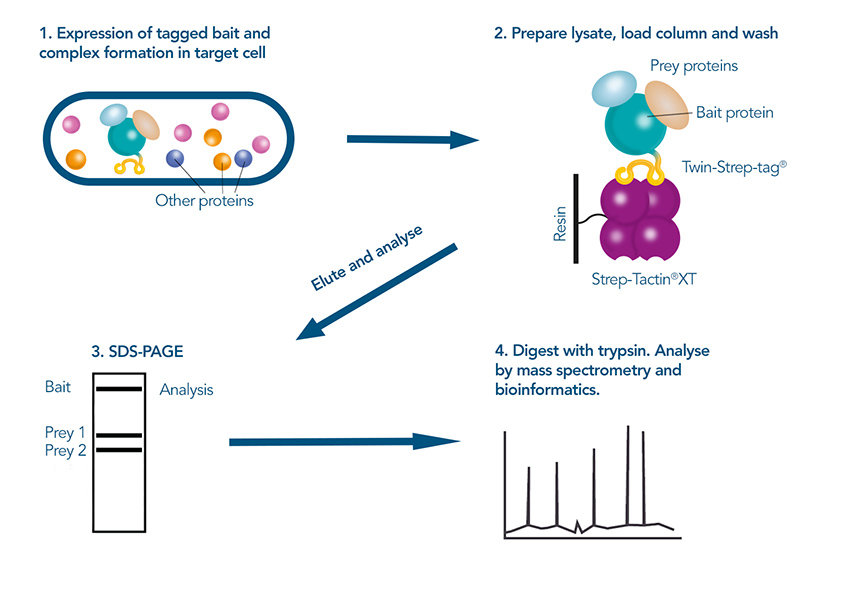
Figure 3: Using a pull-down-assay to identify protein interactions: The tagged target protein is expressed in the host cell, after which it is purified under mild conditions via the Twin-Strep-tag®. Prey proteins that were co purified are separated from the bait protein in gel electrophoresis, digested and then identified in mass spectrometry.
Bestätigung der Interaktion
Nachdem die potenziellen Interaktionspartner identifiziert wurden, sind weitere Schritte erforderlich, um die Interaktion zu bestätigen. Dazu werden das ursprüngliche Bait-Protein und die Prey-Proteine rekombinant mit Modifikationen hergestellt, die den Nachweis ihrer Wechselwirkung erleichtern.
Ein Ansatz besteht darin, die Proteine mit Stoffen zu markieren, die ein Signal erzeugen, wenn sie durch die Interaktion in die Nähe voneinander gebracht werden. Diese Stoffe können Teile von Proteinen oder spezifische Fluorophore sein. Bei homogenen zeitaufgelösten Fluoreszenztests (HTRF) beispielsweise sind Donor- und Akzeptor-Fluorophore mit Molekülen konjugiert, die die Bindungspartner erkennen, wie Strep-Tactin® oder Antikörper. Dieser Assay beruht auf dem Förster-Resonanz-Energie-Transfer (FRET), bei dem Energie von einem Donor-Fluorophor auf ein Akzeptor-Fluorophor übertragen wird und ein Signal erzeugt, wenn sich beide nahe beieinander befinden. HTRF bietet eine hohe Empfindlichkeit und ein geringeres Hintergrundrauschen durch zeitaufgelöste Messung.
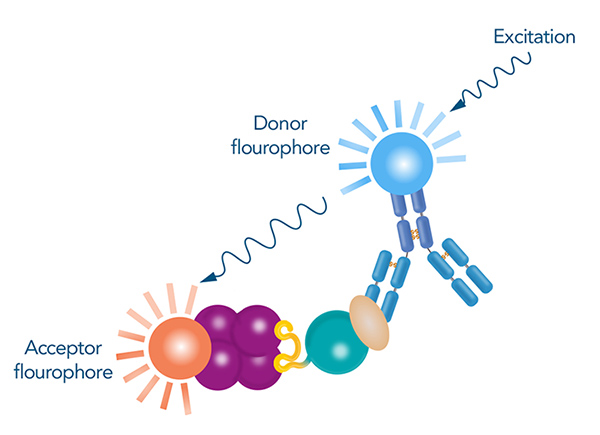
Figure 4: Principle of HTRF for confirmation of protein interactions. The donor fluorophore is conjugated to an antibody specific to the prey protein, while the acceptor fluorophore is conjugated to a ligand that binds to the tag of the bait protein. The donor fluorophore is excited by a light of a certain wavelength and transfers its energy to the acceptor fluorophore if they are in close proximity, which results in a measurable signal.
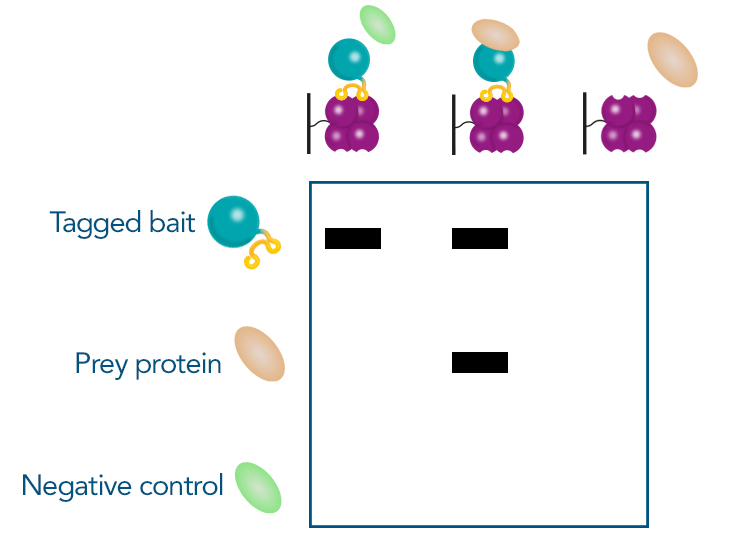
Eine weitere unkomplizierte Methode, insbesondere wenn das erste Screening mit einem Pull-down-Assay durchgeführt wurde, ist ein weiterer anschließender Pull-down. Bei dieser Methode wird das ausgewählte Prey-Protein rekombinant exprimiert und mit dem Bait-Protein gemischt. Anschließend wird eine Affinitätsreinigung für das Bait-Protein durchgeführt. Das Vorhandensein des Prey-Proteins wird durch eine Western-Blot-Analyse bestätigt, wobei die Ergebnisse mit negativen Kontrollen verglichen werden, um die Interaktion zu bestätigen. Andere Methoden bestätigen nicht nur die Interaktion, sondern liefern auch zusätzliche Erkenntnisse. Mit der Proteinkristallographie kann beispielsweise die Proteinstruktur bestimmt werden, und mit der Oberflächenplasmonenresonanz (SPR) lässt sich die Bindungskinetik messen. Diese fortschrittlichen Techniken liefern detaillierte Informationen über die Art und Dynamik der Wechselwirkung.
Figure 5: Confirmation of protein interaction in SDS page after Pull-down-assay. Prior to the experiment, the tagged bait protein, prey protein and an unrelated control protein are recombinantly expressed. The proteins are then mixed in different combinations and purified via the tag: The bait protein with the control protein, the bait protein with the prey protein and the prey protein on its own. If the prey protein is only visible in the gel if it was mixed with the bait protein, the interaction is confirmed.
Charakterisierung der Interaktion: SPR und BLI
Sobald ein Interaktionspartner identifiziert und bestätigt wurde, kann die Interaktion weiter charakterisiert werden, um tiefere Einblicke in ihre biologische Funktion zu gewinnen. Faktoren wie Bindungskinetik und Affinität sind entscheidend für das Verständnis der Rolle der Interaktion im Organismus. Techniken wie Oberflächenplasmonenresonanz (SPR) und Biolayer-Interferometrie (BLI) sind für diesen Zweck besonders nützlich.
Sowohl SPR als auch BLI ermöglichen die Analyse von Proteininteraktionen in Echtzeit und erlauben eine präzise Messung der Assoziations- und Dissoziationsraten. Beide Techniken erfordern die stabile und orientierte Immobilisierung des Köderproteins auf einer Sensor- oder Chipoberfläche. Die Twin-Strep-tag®-Technologie ist für diesen Schritt aufgrund ihrer picomolaren Affinität sehr vorteilhaft, da sie eine sichere und gerichtete Immobilisierung des Köderproteins gewährleistet. Dies ermöglicht die Messung von Wechselwirkungen mit niedriger bis hoher Affinität. Darüber hinaus können mit Strep-Tactin®XT beschichtete Oberflächen regeneriert und mehrfach wiederverwendet werden, was diese Biosensorplattformen kostengünstig macht.
Durch die Nutzung der Stärken von SPR und BLI in Verbindung mit der robusten Immobilisierung durch Affinitäts-Tags wie dem Twin-Strep-tag® können Forscher detaillierte kinetische und Affinitätsdaten erhalten. Diese Informationen sind wichtig, um die funktionelle Rolle von Protein-Protein-Wechselwirkungen und ihre möglichen Auswirkungen auf zelluläre Prozesse und Krankheitsmechanismen aufzudecken.
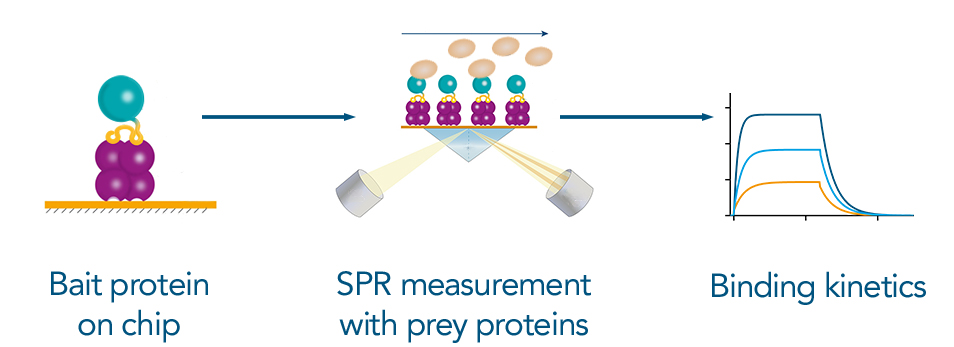
Abbildung 6: Messung der Bindungskinetik mittels SPR: Das markierte Bait-Protein kann über den hochaffinen Twin-Strep-tag® an Strep-Tactin®XT an den SPR-Chip binden. Das Prey-Protein wird zur Messung auf den Chip aufgebracht. Die daraus resultierende Bindungskinetik ist eine wesentliche Information zur Charakterisierung einer Interaktion zwischen zwei Proteinen.
Referenzen
- Gordon, D.E., Jang, G.M., Bouhaddou, M. et al. A SARS-CoV-2 protein interaction map reveals targets for drug repurposing. Nature 583, 459–468 (2020). https://doi.org/10.1038/s41586-020-2286-9
- He L, Valignat MP, Zhang L, Gelard L, Zhang F, Le Guen V, Audebert S, Camoin L, Fossum E, Bogen B, Wang H, Henri S, Roncagalli R, Theodoly O, Liang Y, Malissen M, Malissen B. ARHGAP45 controls naïve T- and B-cell entry into lymph nodes and T-cell progenitor thymus seeding. EMBO Rep. 2021 Apr 7;22(4):e52196. https://doi.org/10.15252/embr.202052196
- Hou YN, Cai Y, Li WH, He WM, Zhao ZY, Zhu WJ, Wang Q, Mai X, Liu J, Lee HC, Stjepanovic G, Zhang H, Zhao YJ. A conformation-specific nanobody targeting the nicotinamide mononucleotide-activated state of SARM1. Nat Commun. 2022 Dec 22;13(1):7898.
https://doi.org/10.1038/s41467-022-35581-y