Proteinreinigung mit magnetischen Beads
Die Proteinreinigung mit magnetischen Beads ist eine hocheffiziente Methode, insbesondere bei kleinen Probenmengen oder viskosen Materialien. MagStrep® Strep-Tactin®XT Beads sind ideal für die Batch-Aufreinigung, da sie eine einfache Trennung vom Überstand und eine hohe Spezifität bieten, was zu außergewöhnlich reinen Proteinen führt. Diese Methode ist vielseitig und skalierbar, eignet sich für unterschiedliche Probengrößen und kann leicht an automatisierte Systeme angepasst werden, was sie perfekt für Hochdurchsatz-Screenings macht. Die mit dem hochaffinen Liganden Strep-Tactin®XT beschichteten Beads ermöglichen eine spezifische Bindung an Strep-getaggte Proteine und bieten einen einfachen und anpassbaren Arbeitsablauf für Ihre experimentellen Anforderungen.
Es ist einfach, die Proteinausbeute zu erhöhen, indem bestimmte Parameter bei der Reinigung von Strep-getaggten Proteinen mit magnetischen Beads angepasst werden. Entdecken Sie diese Optimierungsschritte unten, die Proteingröße, Inkubationszeit, Beadvolumen und Proteinkonzentration beinhalten.
Pull-down-Assays mit magnetischen Beads
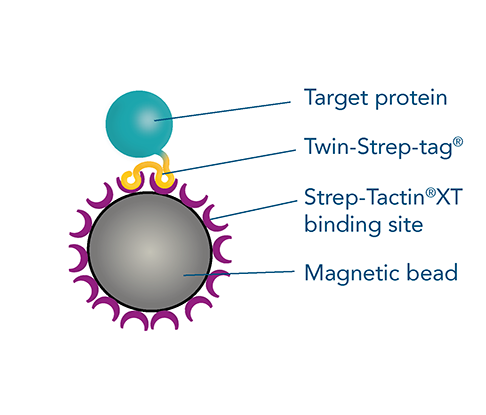
Das Verständnis von Protein-Protein-Wechselwirkungen ist für die Untersuchung von Proteinstruktur und -funktion von entscheidender Bedeutung. Pull-down-Assays sind wesentliche Techniken zum Nachweis dieser Wechselwirkungen, um entweder vorhergesagte Wechselwirkungen zu bestätigen oder neue Wechselwirkungen zu entdecken. Die Pull-Down-Methode ist kostengünstig und bequem. Sie verwendet ein Bait-Protein, das mit einem Affinitäts-Tag, wie dem Twin-Strep-tag®, fusioniert und auf magnetischen Beads immobilisiert ist, die mit dem entsprechenden Liganden, Strep-Tactin®XT, gekoppelt sind.
Erfahren Sie mehr über MagStrep® Strep-Tactin®XT Beads, die eine niedrige pM-Bindungsaffinität mit Twin-Strep-markierten Bait-Proteinen bieten und eine effiziente einstufige Aufreinigung von Komplexen gewährleisten.
Vom frühen Screening bis zur Proteinproduktion im großen Maßstab
Die Aufreinigung auf der Grundlage von magnetischen Beads ist in hohem Maße skalierbar und eignet sich daher für die Verarbeitung einer Vielzahl von Probenmengen. Das Verfahren kann je nach Anwendung an unterschiedliche Probengrößen angepasst werden, von kleinen Hochdurchsatz-Screenings bis hin zur Proteinproduktion im großen Maßstab.
Erfahren Sie mehr über die flexible Skalierung der Proteinreinigung mit magnetischen Beads in dieser Application Note: From Early Screening to Large-Scale Production.
Optimierung der Reinigung von Proteinen auf der Basis von magnetischen Beads
Formular |
5%ige Suspension |
| Bindungskapazität | 42,5 mg/ml (0,85 nmol/µl eines 50 kDa-Proteins) |
| Matrix | 6% Agarose, vernetzt, kugelförmige magnetische Beads |
| Durchmesser der Beads | 30 µm durchschnittlich |
| Spezifität | Strep-tag®II und Twin-Strep-tag® |
Optimierungsparameter
Das IBA-Protokoll für MagStrep® Strep-Tactin®XT Beads beschreibt die grundlegenden Schritte für die magnetische Reinigung von Proteinen. Abhängig von Ihren experimentellen Bedingungen ist es möglich, das Reinigungsprotokoll zu optimieren und die Proteinausbeute zu erhöhen, indem bestimmte Parameter angepasst werden. Die folgenden Tipps und Daten helfen Ihnen bei der Optimierung der Magnetbead-Proteinreinigung durch Anpassung von Beadvolumen, Inkubationszeit und Proteinkonzentration.
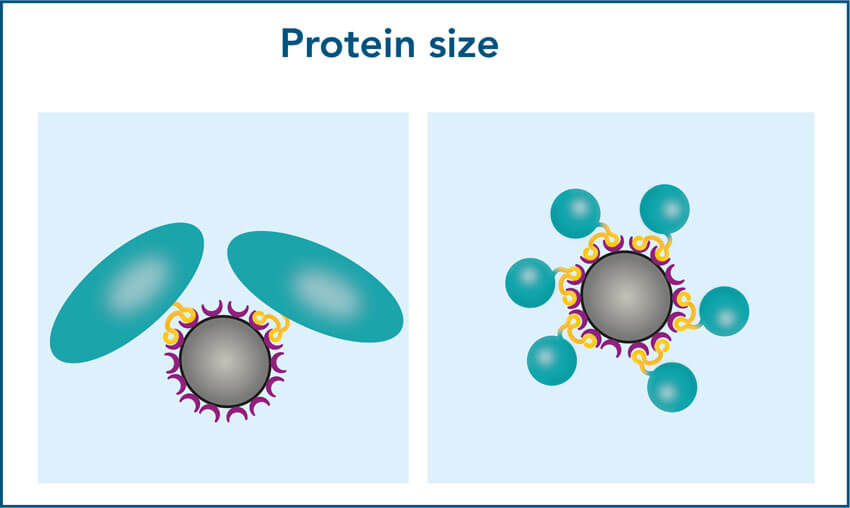
1. Proteingröße
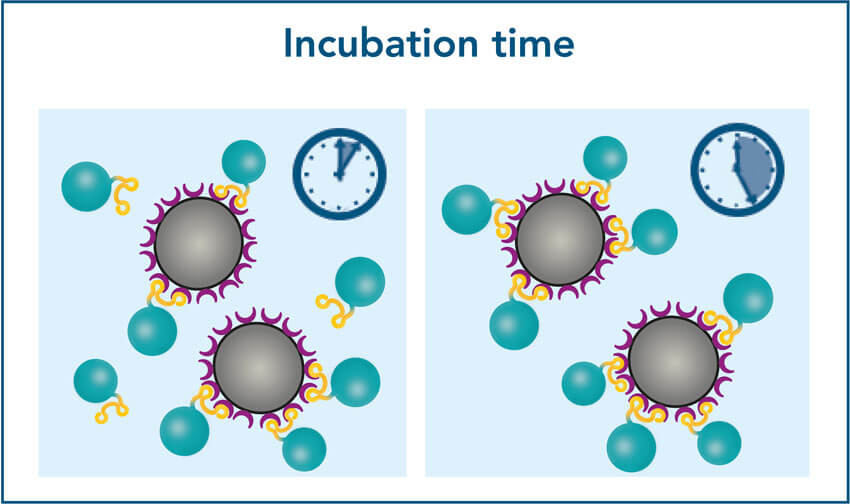
2. Inkubationszeit
1. Das erforderliche Beadvolumen hängt von der Größe des Proteins ab
Bei der Arbeit mit kleinen Proteinen werden mehr Moleküle pro Bead gebunden als bei großen Proteinen. Aufgrund ihrer größeren Oberfläche benötigen große Proteine mehr Platz zum Binden. Infolgedessen kann die Bindekapazität für Proteine >90 kDa merklich abnehmen, was zu einer geringeren Ausbeute führen kann.
Um eine hohe Ausbeute bei großen Proteinen zu gewährleisten, erhöhen Sie das Beadvolumen, um eine ausreichende Bindeoberfläche zu schaffen. Das optimale Beadvolumen für Ihr spezifisches Protein kann durch Titration bestimmt werden.
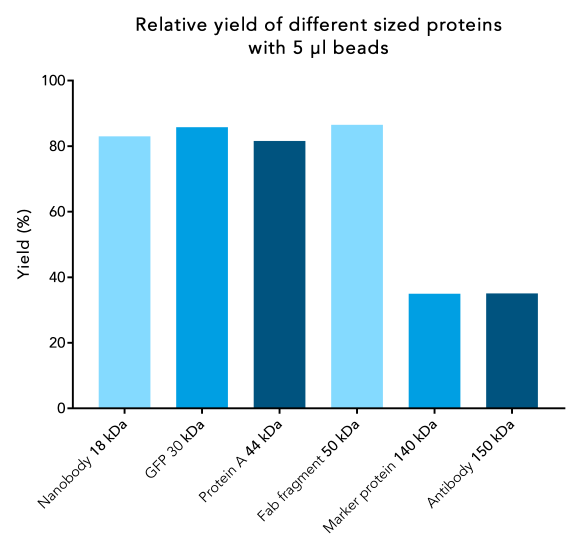
Abbildung 1: Die Proteinausbeute nimmt bei großen Proteinen ab. 250 µl von Twin-Strep-tag® Fusionsproteinen unterschiedlicher Größe mit einer Ausgangsproteinkonzentration von 1,7 nmol/µl wurden mit 5 µl Beads inkubiert. Die Proteinreinigung erfolgte nach dem IBA-Standardprotokoll. Nach der Elution wurde der Gesamtproteingehalt mit dem Proteingehalt zu Beginn verglichen.
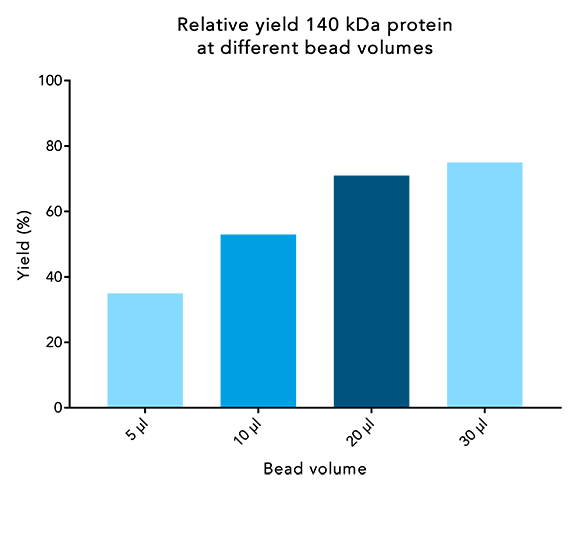
Abbildung 2: Die Proteinausbeute bei großen Proteinen steigt, wenn höhere Beadvolumina verwendet werden. 250 µl eines 140 kDa-Proteins mit 1,7 nmol/µl wurden 10 Minuten lang mit verschiedenen Bead-Volumina inkubiert. Anschließend wurde die Reinigung nach dem IBA-Standardprotokoll durchgeführt. Der Proteingehalt der Elution wurde mit dem Ausgangsproteingehalt verglichen.
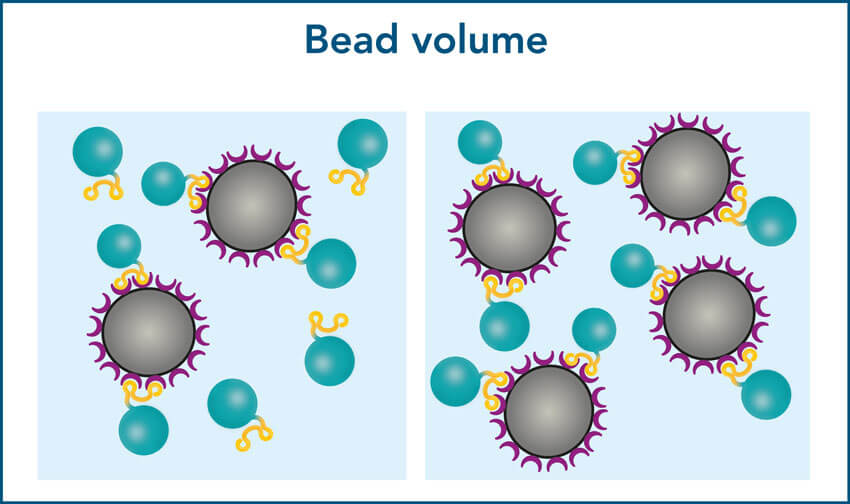
2. Erhöhte Beadvolumina verkürzen die Inkubationszeit
Um eine höhere Proteinausbeute bei kürzerer Inkubationszeit zu erreichen, fügen Sie einen Überschuss an Beads im Verhältnis zur Proteinmenge und Bindekapazität der Beads hinzu (0,85 nmol/µl Beads oder 42,5 µg/µl eines 50 kDa-Proteins). Die beste Ausbeute im Verhältnis zum verwendeten Beadvolumen wird erreicht, wenn 5x mehr Beads im Verhältnis zur maximalen Bindekapazität hinzugefügt werden. Beträgt die Gesamtproteinmenge z. B. 85 μg, die theoretisch von 2 μl Beads gebunden werden könnte, werden 5x 2 μl = 10 μl Beads zugegeben und 10 Minuten lang inkubiert, um die beste Ausbeute zu erzielen.
Die maximale Ausbeute kann auch bei Verwendung eines geringeren Bead-Volumens erreicht werden, aber die Inkubationszeit kann deutlich länger sein.
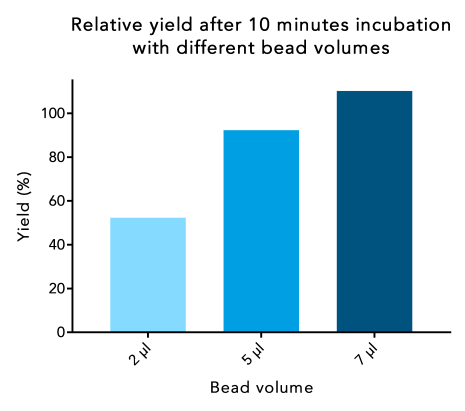
Abbildung 3: Bei Verwendung eines höheren Beadvolumens kann die Proteinausbeute bei kurzer Inkubationszeit erhöht werden. 250 µl eines 30 kDa Twin-Strep-tag® Fusionsproteins wurden mit verschiedenen Beadvolumina für die gleiche Inkubationszeit inkubiert. Der Proteingehalt der Elution wurde mit dem Proteingehalt zu Beginn verglichen.
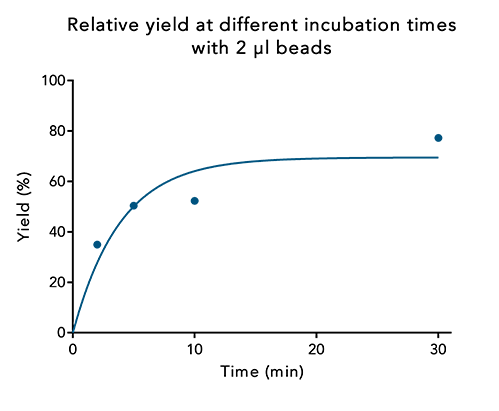
Abbildung 4: Bei einem niedrigen Beadvolumen kann die Proteinausbeute durch eine längere Inkubationszeit erhöht werden. 250 µl eines 30 kDa Twin-Strep-tag® Fusionsproteins wurden mit demselben Beadvolumen für verschiedene Zeiträume inkubiert. Der Proteingehalt der Elution wurde mit dem Proteingehalt zu Beginn verglichen.
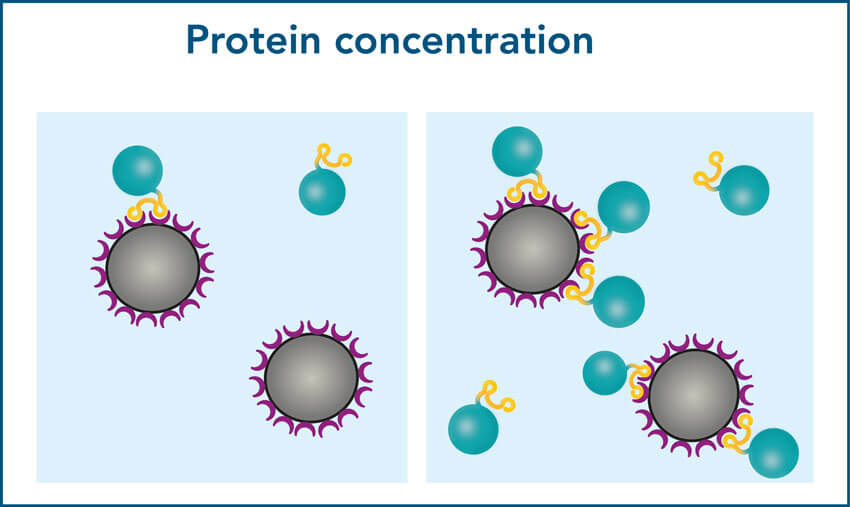
3. Probenproteinkonzentration und Beadvolumen müssen ausgeglichen sein
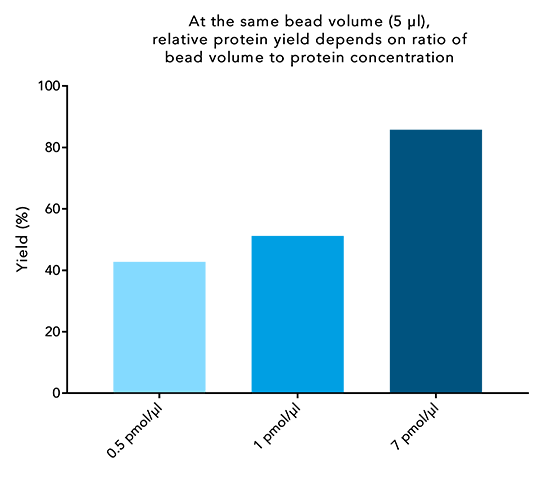
Abbildung 5: Höhere Proteinkonzentrationen führen zu einer höheren Proteinausbeute. 250 µl verschieden konzentrierter Proben eines 30 kDa Twin-Strep-tag® Fusionsproteins wurden mit 5 µl Beads 10 Minuten lang inkubiert. Anschließend wurde das IBA-Standardprotokoll durchgeführt und der endgültige Proteingehalt der Elution mit dem Proteingehalt zu Beginn verglichen.
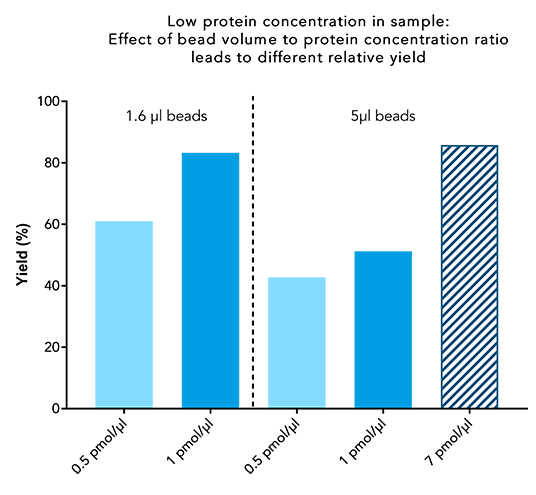
Abbildung 6: Ein hohes Beadvolumen führt nicht zu einer hohen Ausbeute, wenn mit niedrigen Proteinkonzentrationen gearbeitet wird. 250 µl Probe, die 0,5 oder 1 pmol/µl eines 30 kDa Twin-Strep-tag® Fusionsproteins enthalten, wurden 10 Minuten lang mit 1,6 oder 5 µl Beads inkubiert. Anschließend wurde das IBA-Standardprotokoll durchgeführt und der endgültige Proteingehalt der Elution mit dem Proteingehalt zu Beginn verglichen.
FAQ zur Magnetic-Bead-Reinigung
Anwendungsbeispiel
GFP C-terminal getaggt mit Strep-tag®II (28 kDa) wurde in E. coli exprimiert (A) und mit MagStrep® Strep-Tactin®XT Beads (MagStrep "type3" XT Beads) gereinigt. Zur Abtrennung der magnetischen Beads aus der Probe wurde der Magnetic Separator verwendet. Vor der Elution wurde die Probe geteilt und das Zielprotein entweder durch Auftragen von 1x Buffer BXT mit Biotin oder durch Kochen (C, 5 min bei 95 °C) eluiert. Durch das Kochen wird die Agarose des Beads zerstört und Strep-Tactin®XT wird freigesetzt, was zu einem weiteren Peak bei 14 kDa führt. Die Ergebnisse der Proteinreinigung wurden mit dem Agilent Bioanalyzer 2100 System analysiert. Das Beispiel zeigt die spezifischen Bindeeigenschaften von MagStrep® Strep-Tactin®XT Beads (MagStrep "Typ3" XT Beads) und die dadurch zu beobachtende hohe Reinheit.