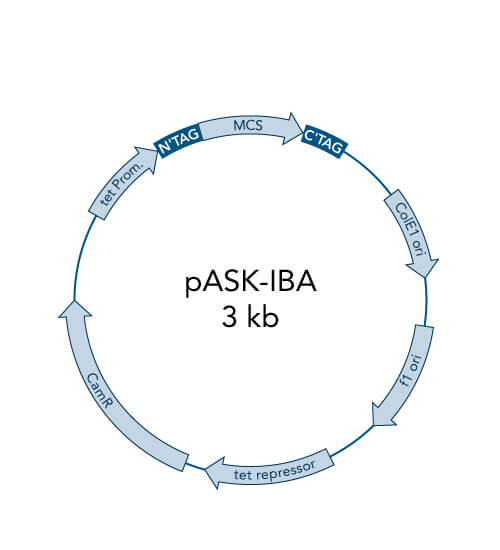
Klonieren
Ziel der molekularen Klonierungsverfahren ist es, ein gewünschtes Gen in einen geeigneten Expressionswirt einer kompatiblen Weise (rekombinante DNA) einzubringen, um schließlich das Genprodukt (Protein) in zufriedenstellender Qualität und Quantität herzustellen. Klonen bedeutet die Herstellung identischer Kopien eines bestimmten Gens von Interesse. Bei der Klonierung wird das Gen vervielfältigt und dann in ein Plasmid (Vektor) eingefügt, um es anschließend zu replizieren und das Protein zu exprimieren. Die Verwendung von Plasmiden ermöglicht den Schutz des fremden genetischen Materials vor den angeborenen Abbaumechanismen des Expressionswirts.
Es gibt verschiedene Klonierungsverfahren, um das gewünschte Gen in ein Plasmid einzufügen. Das Klonieren auf der Basis von Restriktionsenzymen ist die Standardklonierungsmethode in der Molekularbiologie. Restriktionsendonukleasen spalten doppelsträngige DNA (dsDNA) an spezifischen Sequenzstellen, den sogenannten Erkennungsstellen. Je nach verwendetem Restriktionsenzym können die erzeugten DNA-Fragmente entweder stumpfe Enden oder klebrige Enden haben. Diese können mit Plasmid-DNA fusioniert werden, wenn diese mit demselben Restriktionsenzym gespalten und dadurch linearisiert vorliegt. Die Fusionierung des DNA-Fragments und der linearisierten Plasmid-DNA erfolgt mit Hilfe einer sogenannten DNA-Ligase.
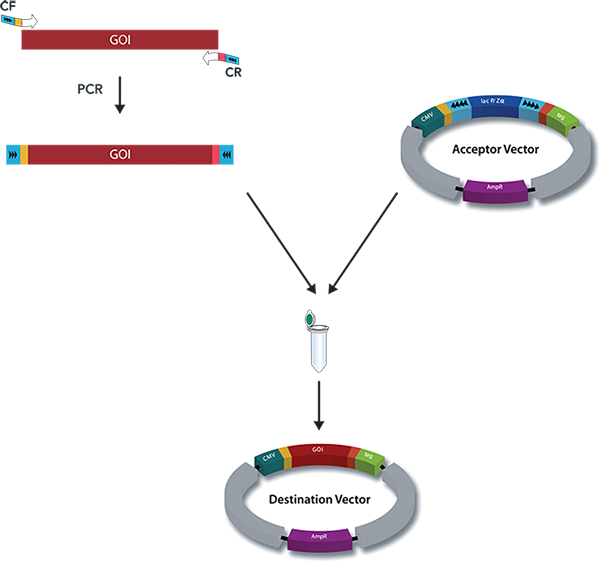
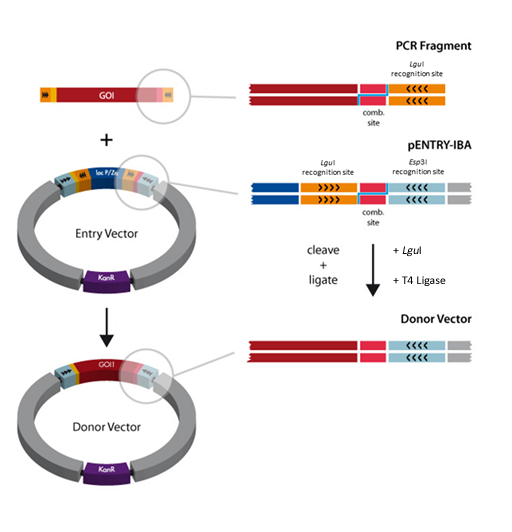
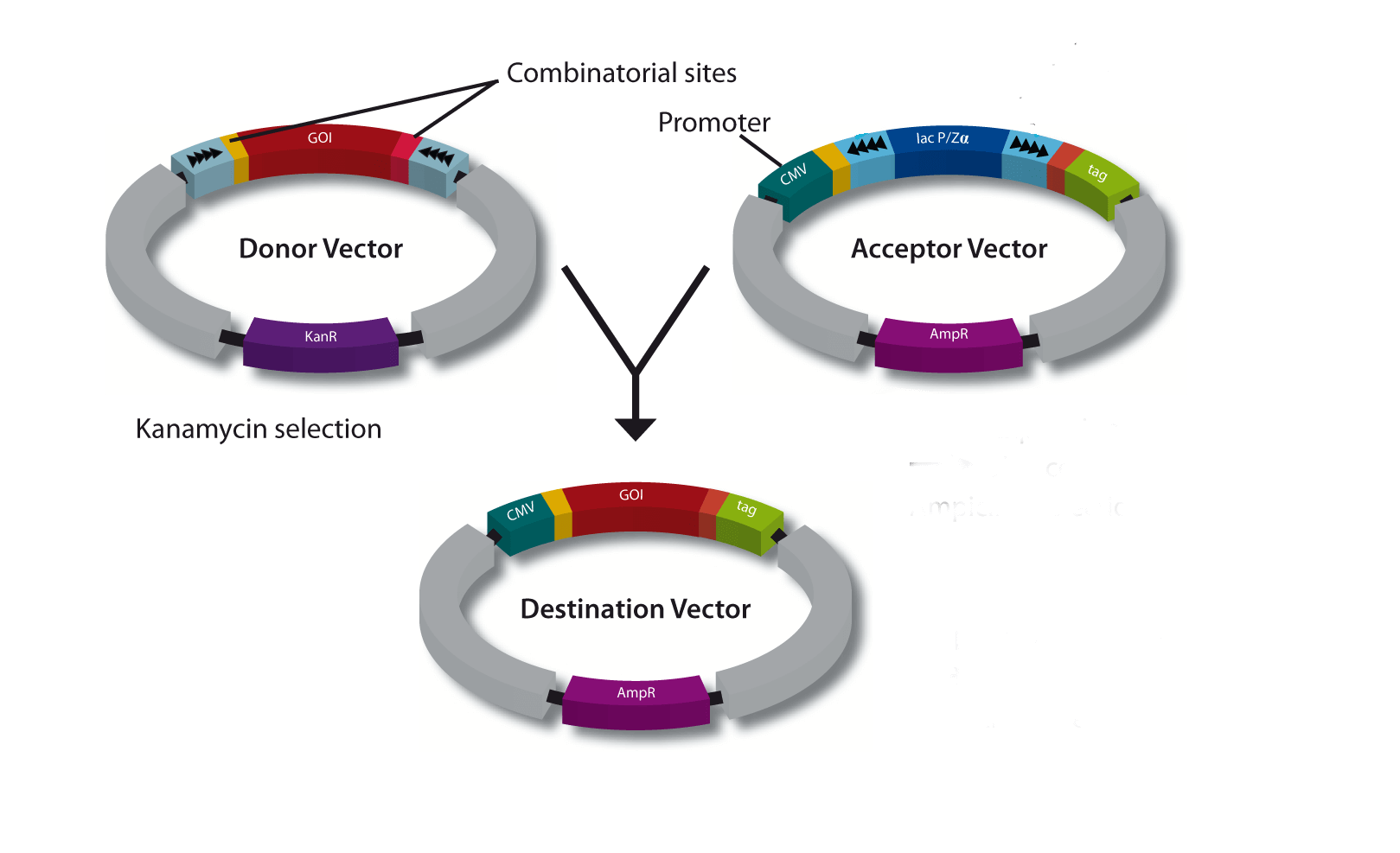
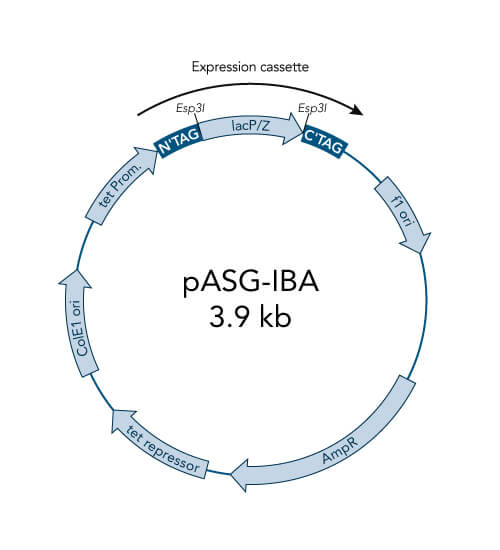
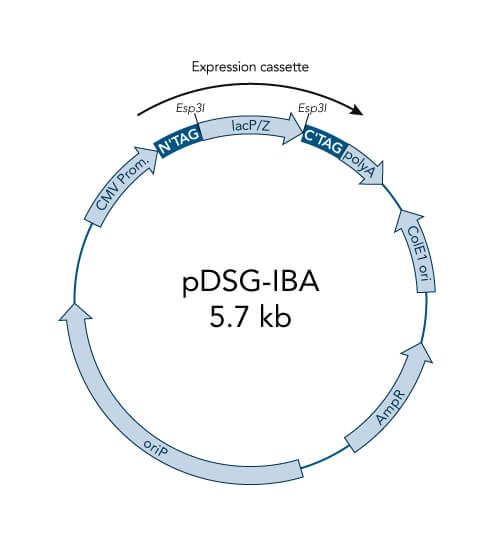
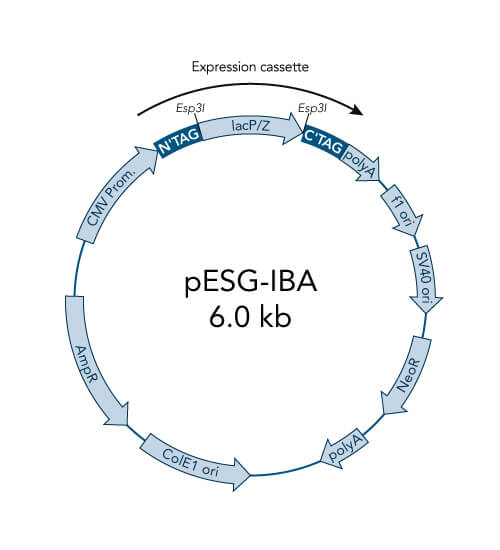
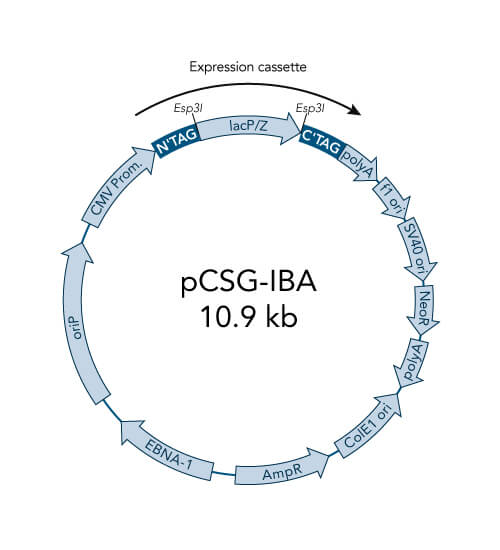
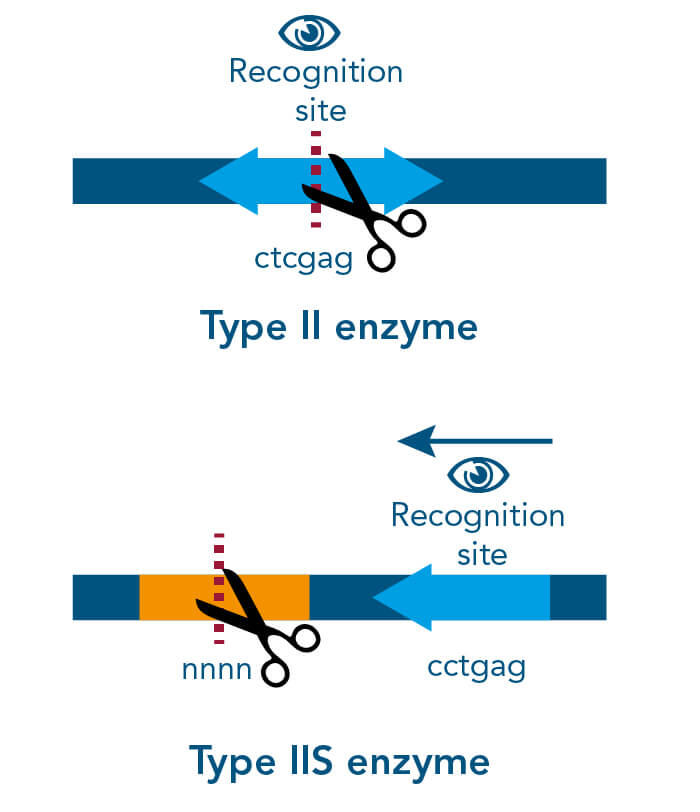
Eine spezielle Klonierungsmethode nutzt die spezifischen Eigenschaften von Typ-IIS-Restriktionsenzymen, Endonukleasen, die DNA außerhalb der Erkennungssequenz schneiden. Im Gegensatz zur traditionellen Klonierung mit Restriktionsenzymen bietet dieses Schneiden außerhalb der Erkennungsstelle den Vorteil, dass individuelle Überhänge erzeugt werden können. Diesen Ansatz hat IBA Lifesciences auch für das StarGate-System genutzt, um kompatible individuelle Überhänge zu erzeugen, die dann effizient fusioniert werden können. Das StarGate-System bietet eine große Anzahl an Klonierungs- und Expressionsvektoren für E. coli und Säugetierzellen.
StarGate für schnelles Klonieren
StarGate wurde für das schnelle systematische Screening des optimalen Expressions- (in Bezug auf den Expressionswirt und/oder den Promotor) und Reinigungssystems (in Bezug auf den Affinitäts-Tag) für ein bestimmtes Gen von Interesse (GOI) entwickelt.